Proyectos de clase
Experimento 2018-S2
Este experimento fue realizado por los estudiantes de ecotoxicología del semestre 2018-2. Se sometieron plántulas de lechuga a distintas dosis de fertilizante diluidas en masas de tierra. Para analizar los efectos del fertilizante en la lechuga se utilizo un diseño experimental Dosis respuesta analizado con un análisis de varianza (ANOVA).
- Condiciones para Anova
- Normalidad de los residuos.
- Independencia de los residuos.
- Homocedasticidad de los residuos.
Hipótesis anova \[H_0 : \mu_1\; =\; \mu_2\; =\; \mu_3\; =\; ...\mu_n\; \equiv\; \] \[H_0 : \forall_i\; : \mu_i\; = \mu_\;\] \[H_1 : Al\;menos\;una\;media\;difiere\;de\;las\;demas\] \[\alpha =\; 0,05\]
- Descomposición de la varianza (\(\sigma^{2}\)):
- Variabilidad total : Suma de cuadrados total (SCT) : Variabilidad entre grupos + Variabilidad dentro de grupos .
- Variabilidad del tratamiento (intervarianza) : Suma de cuadrados entre grupos.
- Variabilidad del error (intravarianza) : Suma de cuadrados residual o dentro de grupos.
Valor F:
El valor F mide el grado de similitud que existe entre las medias que se están comparando. El valor F aumenta a mayor diferencia en las medias
- \(F \approx 1\) : Medias iguales
- \(F > 1\) : Medias distintas
\[F =\; \frac{Suma\; de\; cuadrados\;entre\;grupos}{Suma\; de\; cuadrados\;dentro\;de\;grupos}\;\;\ \equiv\; \frac{Variabilidad\; del\; Tratamiento\;}{Variabilidad\; del\;Error} \]
Tamaño del efecto (\(\eta^{2}\)):
El tamaño del efecto de una ANOVA permite cuantificar cuánto afecta la variable independiente (factor) a la variable dependiente.
- \(\eta^{2} \leq 0,05\) : Efecto pequeño o debil
- \(0,05 < \eta^{2} \leq 0,14\) : Efecto mediano o moderado
- \(\eta^{2} > 0,14\) : Efecto grande o fuerte
\[\eta^{2} =\; \frac{Suma\; de\; cuadrados\;entre\;grupos}{Suma\; de\; cuadrados\;total\;}\;\;\ \equiv\; \frac{Variabilidad\; del\; Tratamiento\;}{Variabilidad\; Total\;}\]
Datos
Gráfico exploratorio
g <- ggplot(data = df, aes(x = Dosis, y = Area)) + theme_minimal()+
geom_jitter(width = 0.1, height = 0,size=2,alpha=0.2,shape=19) +
expand_limits(y=c(0,100))+
stat_summary(fun.y = "mean", geom = "pointrange", col = "#E69F00",
shape = 19, size=0.7, fun.ymin = function(x) mean(x) - sd(x),fun.ymax = function(x) mean(x) + sd(x)) +
stat_summary(fun.y = "mean", geom = "errorbar", width=0.2,lwd=1, col = "#E69F00",fun.ymin = function(x) mean(x) - sd(x),fun.ymax = function(x) mean(x) + sd(x)) +
stat_summary(fun.y = "mean", geom = "line", col = "#E69F00", group = 1,lwd=0.7) +
geom_hline(yintercept = mean(df$Area), lwd = 1, color = "#56B4E9",
lty = 2) +
geom_text(mapping=aes(x=5.2,y=13),label="",vjust=-0.3,color="blue",size=2) +
labs(x = "", y = "Area(cm2)",title = "Lactuca sativa")
#labs(x = "", y = expression('Area '*('cm'^'2')),title = "Lactuca sativa")
ggplotly(g)Modelo Anova
modelo<-aov(data = df,formula = Area~Dosis )
summary(modelo)## Df Sum Sq Mean Sq F value Pr(>F)
## Dosis 4 25751 6438 114.3 <2e-16 ***
## Residuals 94 5296 56
## ---
## Signif. codes: 0 '***' 0.001 '**' 0.01 '*' 0.05 '.' 0.1 ' ' 1Medias
model.tables(x = modelo, type = "means")## Tables of means
## Grand mean
##
## 24.0903
##
## Dosis
## Control Dosis 1 Dosis 2 Dosis 3 Dosis 4
## 52.3 29.72 19.3 13.24 6.174
## rep 20.0 19.00 20.0 20.00 20.000Efectos
model.tables(x = modelo, type = "effects")## Tables of effects
##
## Dosis
## Control Dosis 1 Dosis 2 Dosis 3 Dosis 4
## 28.21 5.633 -4.791 -10.85 -17.92
## rep 20.00 19.000 20.000 20.00 20.00Residuales y supuestos
#Residuales estandarizados
resid.estandar <- rstandard(model = modelo)
#Agregando residuales a la base de datos
df$Residuales <- resid.estandarNormalidad
# Gráfico Q-Q
car::qqPlot(df$Residuales, main = "Residuales del modelo",id=FALSE,ylab = "res")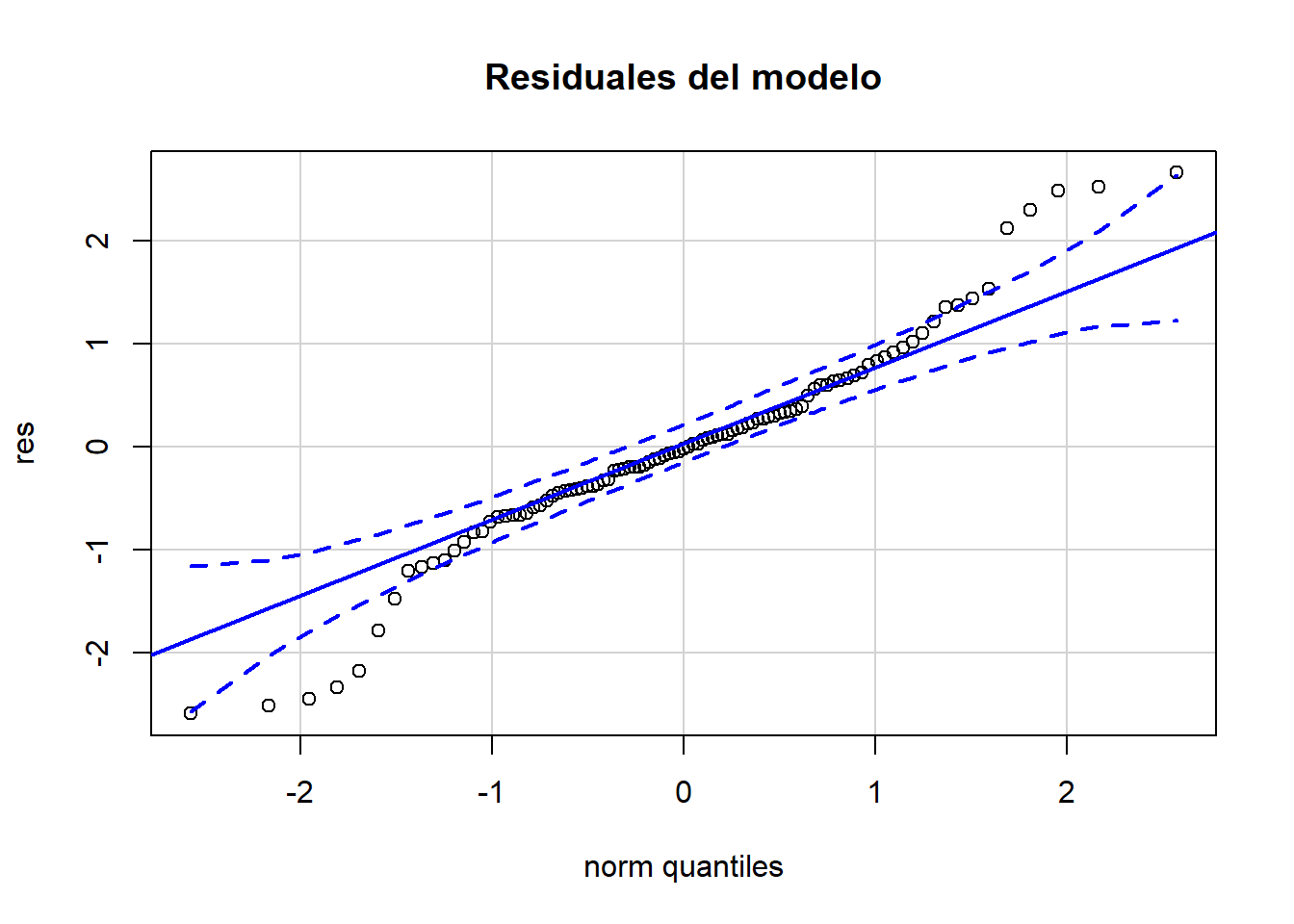
#Prueba de normalidad
shapiro.test(df$Residuales)##
## Shapiro-Wilk normality test
##
## data: df$Residuales
## W = 0.96226, p-value = 0.006191Homogenidad de varianzas
bartlett.test(df$Residuales ~ df$Dosis)##
## Bartlett test of homogeneity of variances
##
## data: df$Residuales by df$Dosis
## Bartlett's K-squared = 45.509, df = 4, p-value = 3.115e-09Tamaño del efecto
lsr::etaSquared(modelo)## eta.sq eta.sq.part
## Dosis 0.8294305 0.8294305